
复旦大学附属华山医院神经外科吴劲松教授、金雷博士、病理科陈宏副主任医师团队在周良辅院士、毛颖院长的指导下携手联影智能沈定刚教授团队在人工智能神经肿瘤病理辅助诊断的工作中取得突破,相关论文成果Artificial Intelligence Neuropathologist for Glioma Classification using Deep Learning on Hematoxylin and Eosin Stained Slide images and Molecular Markers于上月在神经肿瘤领域国际期刊《Neuro-Oncology》(影响因子10.246)上发表。
图一:原文已在Neuro-oncology期刊网站上线
病理是脑胶质瘤诊断的金标准,世界卫生组织WHO中枢神经系统肿瘤分类指南(2016)中将分子信息纳入了脑胶质瘤病理诊断的标准中。通常,病理诊断需通过细胞层面的组织H&E染色切片学进行观察,结合免疫组化进行诊断病理医生诊断所需时间较长,且存在一定的主观性,可能导致诊断不一致的情况出现。基层病理资源尤为匮乏,较难开展后续诊疗。因此,研究者提出了借助AI提高胶质瘤病理诊断的效率与精度的设想。
该文章中介绍了研究者以华山医院神经外科中枢神经系统疾病生物样本库的大量神经病理数据为基础,探索AI深度学习技术在组织病理图像识别中的应用方法,根据多样的深度学习算法的尝试与革新,研发出了一套最适合脑胶质瘤病理多分类的算法:加权的压缩激励密集深度学习网络(SD-Net-WCE)。
在开发诊断模型的同时,研究者同时研发了与之匹配的微动显微平台:通过加装工业摄像头和微动平台,即可将传统的显微镜改造为数字扫描显微镜,从而可低成本地实现病理切片数字化。通过设定的程序,微动显微平台可自动以“之”字形顺序扫描病理切片,完成400倍成像,图片之间留有部分重叠区域。在AI加持之下,算法首先对每张图片的镜头畸变和不均匀光场进行矫正,相邻图像之间通过形态学算子找到对应点,从而可将整张病理切片的大图拼接起来。该方法可实现病理切片数字化,有助于解决病理切片的物理空间占用问题及存储时可能出现的损坏等情况,促进病理数据共享。
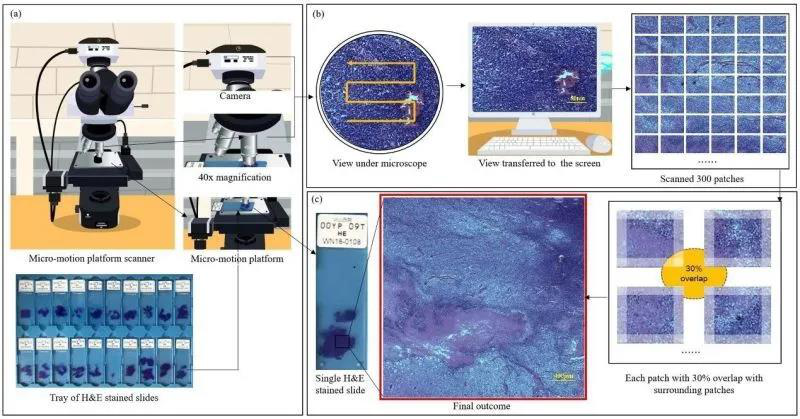
图二:微动显微平台与图像拼接技术
基于病理切片拼接图像,输入的扫描图像可通过加权的压缩激励密集深度学习网络得出图像级的胶质瘤类型的概率,根据拼接图像上的概率分布、运用决策树可获取病例级结果。在五折交叉验证的结果表明,图像级的诊断精准度达到86.5%,而病例级的诊断精度达到了87.5%。该模型可完成以下组织病理胶质瘤分类分级:少突胶质细胞瘤(Ⅱ级)和间变少突胶质细胞瘤(Ⅲ级),星形细胞瘤包括星形细胞瘤(Ⅱ级)、间变性星形细胞瘤(Ⅲ级)和胶质母细胞瘤(Ⅳ级)。
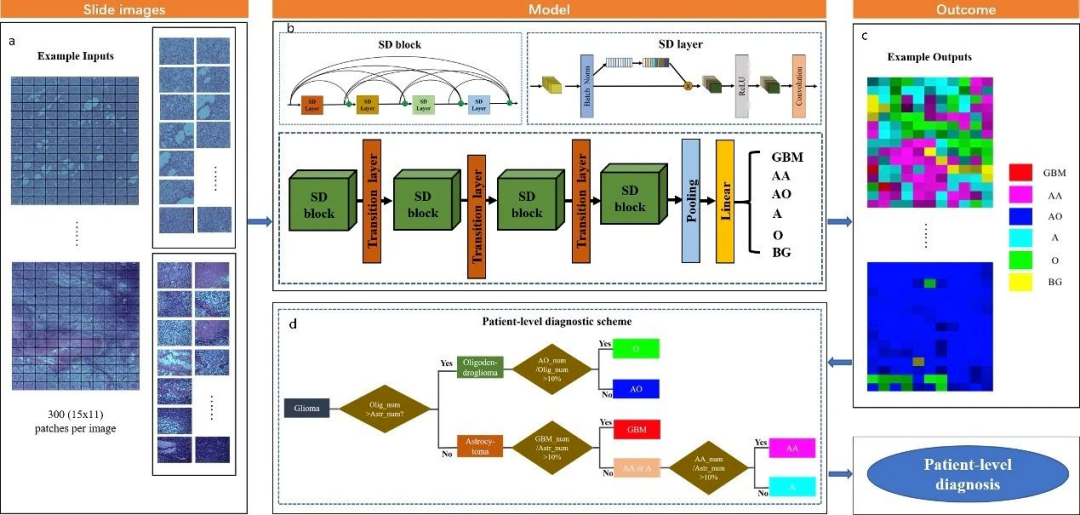
图三:脑胶质瘤的组织病理图像分类分级方法模型
最终,系统可将WHO2016版指南中分子病理信息IDH与1p/19q整合到模型中,从而依据指南获取最终的整合病理诊断结果。由此,本文的方法可辅助诊断脑胶质瘤并进行分类分级,有效弥补人工诊断效率低、缺乏统一的质控管理等问题。在一定程度上可缓解病理医生短缺、供需不均及分布不均等现状。
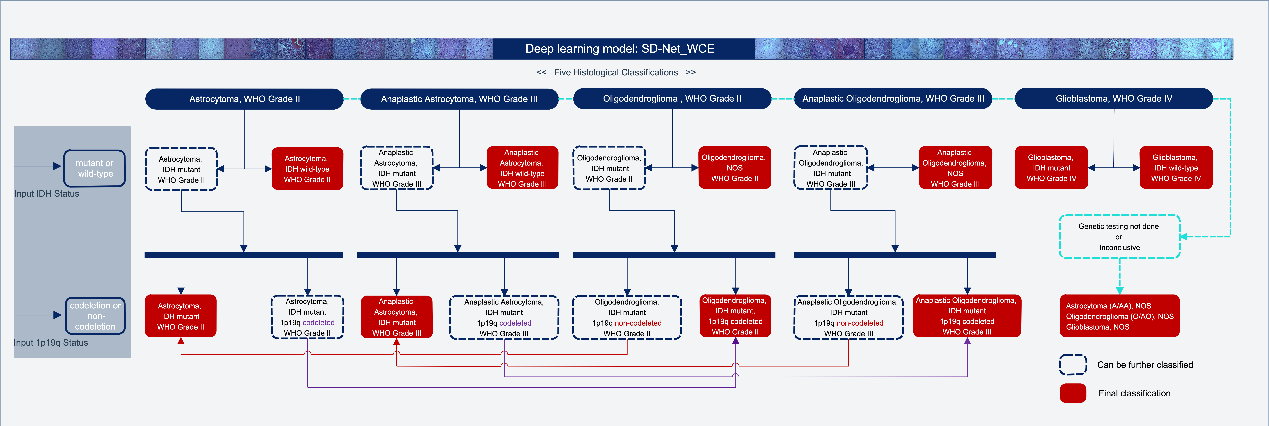
图四:分子病理逻辑推导图
华山医院金雷博士和联影智能石峰博士为本文共同第一作者,华山医院吴劲松教授和联影智能沈定刚教授为本文共同通讯作者;同时感谢中纪生物的技术支持。
参考文献
Lei Jin#, Feng Shi#, Qiuping Chun, Hong Chen, Yixin Ma, N U Frarrukh Hameed, Shuai Wu, Chunming Mei, Junfeng Lu, Jun Zhang, Abudumijiti Aibaidula, Dinggang Shen*, Jinsong Wu*, Artificial Intelligence Neuropathologist for Glioma Classification using Deep Learning on Hematoxylin and Eosin Stained Slide images and Molecular Markers, Neuro-Oncology, noaa163, https://doi.org/10.1093/neuonc/noaa163




