最近,在国际著名期刊ActaNeuropathologica上发表了名为“Routine RNA sequencing offormalin‑fixed paraffin‑embeddedspecimens in neuropathology diagnostics identifies diagnostically andtherapeutically relevant gene fusions”的报道,评估了在常规诊断环境中通过下一代mRNA测序研究FFPE肿瘤组织中基因融合的可行性和临床价值。
研究背景
2016年中枢神经系统WHO分类首次推出了结合组织分型与分子分型的肿瘤分类新标准。许多与脑肿瘤预后相关和与用药物相关靶点的基因被建议进行检测。IDH1R132H、BRAFV600E和H3F3AK27M等点突变可用免疫组化或一代测序进行检测;1p/19q、EGFR和PTEN的拷贝数变异突变可用FISH、MLPA和基于DNA的NGS检测。然而,在脑肿瘤中存在的大量的融合突变,例如BRAF、MYB、MYBL、FGFR、EGFR、RELA和NTRK等基因的融合。利用上述的检测技术仅仅只能部分覆盖驱动脑肿瘤形成的融合突变。在常规分子诊断工作中只能不重复或仅有选择性地评估基因的重排融合,而RNA测序能涵盖所有的结构畸变。对整个mRNA的测序甚至可以检测罕见融合事件,而不受限于预定义融合子集方案的限制。基于以上背景,作者评估了在常规诊断环境中通过下一代mRNA测序研究FFPE肿瘤组织中基因融合的可行性和临床价值。
研究方法
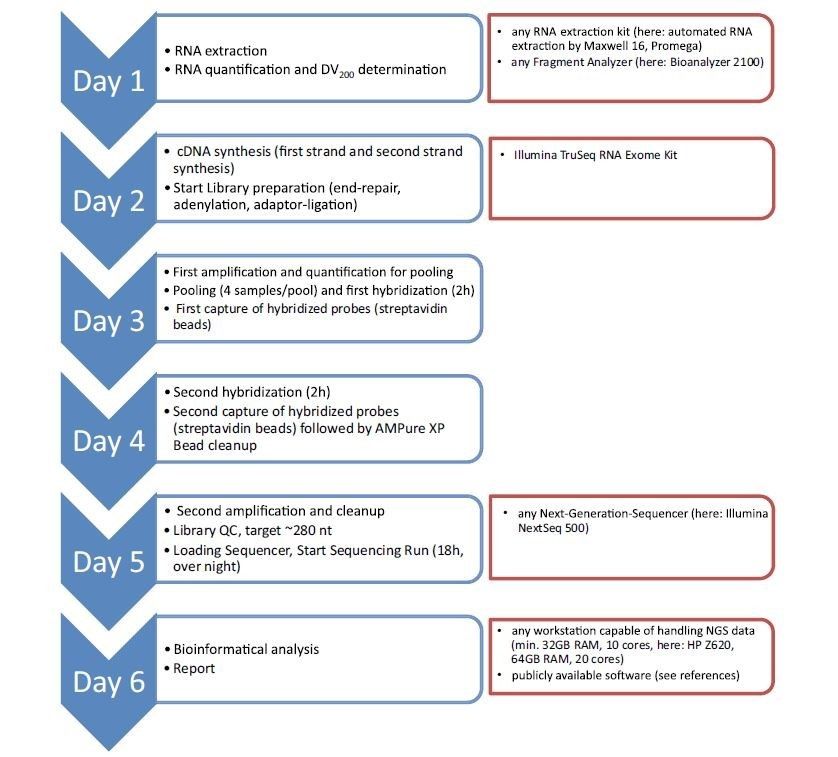
该研究首先从样本库中选择了符合条件的入组肿瘤样本,对其进行了DNA甲基化分析、DNA的panel测序以及免疫组化检测;然后根据图1中的实验诊断流程对入组样本进行RNA测序;最后对RNA测序后的数据进行相关的生物信息学分析。
图1:神经病理学诊断中RNA测序的实验流程:方案步骤和涉及的设备的流程图
研究结果
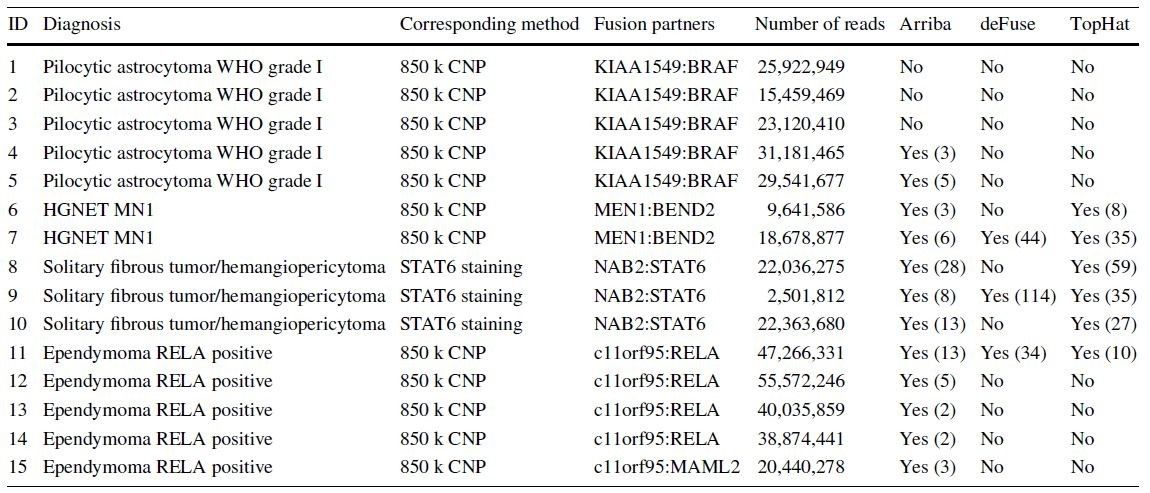
该研究使用了公开的数据分析工具deFuse,TopHat和Arriba用于分析RNA测序数据中的融合突变,用15例已用别的方法检测过融合突变的样本进行新开发的mRNA NGS测序和分析,经过结果比对证明了可以从FFPE RNA测序数据中检测诊断相关的基因融合。在三种测试算法中,Arriba成为最可靠和最有效的工具,在验证实验中达到了80%的灵敏度(表1)。
表1:用于验证RNA-NGS检测融合方法的15例脑肿瘤相关信息
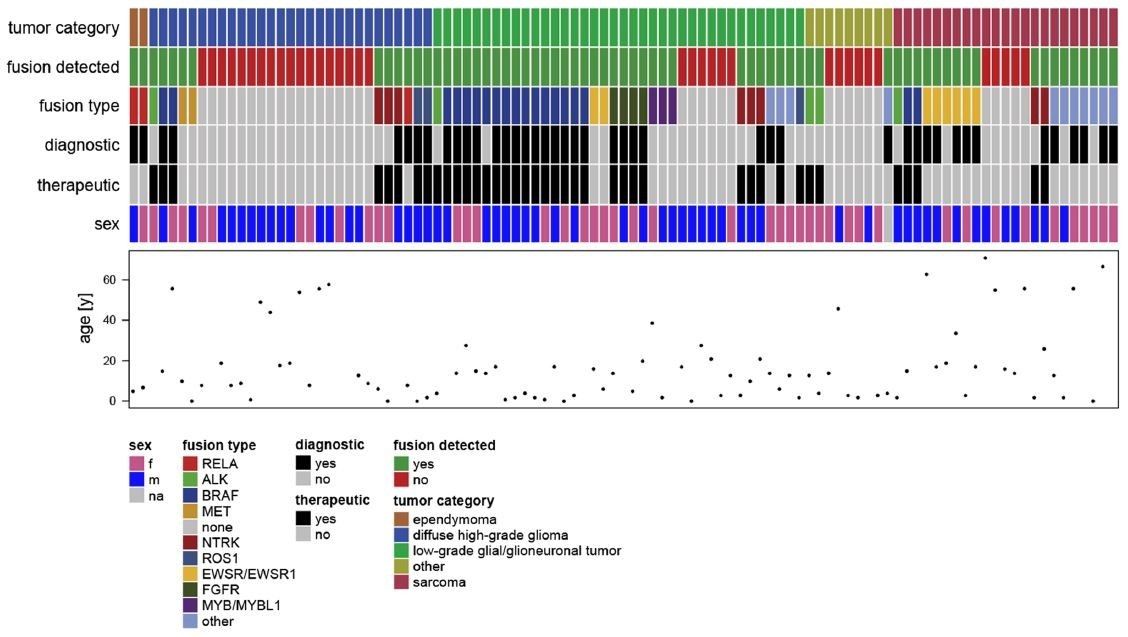
之后,利用上述方法对101例脑肿瘤样本进行了测序,分析其融合突变的情况。结果显示在n = 66/101例中检测到相关融合,低级别的脑肿瘤中携带有融合突变的概率大,在这些低级别的脑肿瘤利用DNA的panel和850K芯片测序时没有发现常见的融合突变(BRAF和FGFR1),可能的原因是DNA的panel和850K芯片测序存在基因断点。39.6%(40/101)样本中检测出来的与药物相关的融合突变,大部分是MAPK通路的靶点,此外还有一些融合是NTRK1/2/3、ROS/ALK和FEFR也被检测到(图2)。
图2:前瞻性群组信息:101样本的特征包括诊断病例和确定的融合。
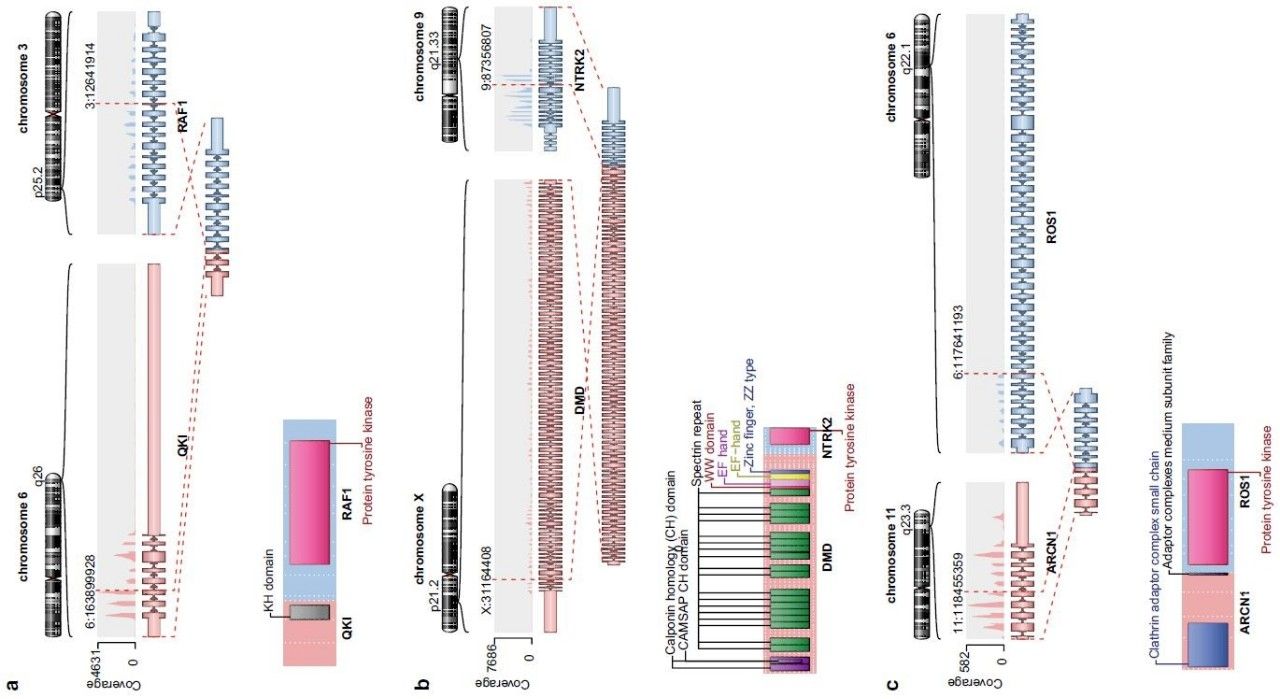
该研究最后分享三例用850K和DNA panel测序是阴性,但用RNA NGS检测是阳性的融合突变案例。第一个案例是被诊断为WHO II级的毛细胞黏液星形细胞瘤,用RNA NGS检测出其携带有QKI:RAF1的融合;第二个案例是被诊断为WHO I级的毛细胞性星形细胞瘤,检测出具有DMD:NTRK2的融合;最后一个案例是被诊断为WHO IV GBM的儿童高级别胶质瘤,该患者利用RNA NGS测序检测出了ARCN1:ROS1的融合(图3)。
图3:三个案例患者中携带的融合突变方式
结论
总之,该研究证明了全面的mRNA测序方法可以常规应用于诊断FFPE样品。基于mRNA的NGS测序分析可以提供对其他诊断上无法解决的病例的生物学的实质性洞察,并立即识别潜在用药的相关基因融合信息。
文章来源:Acta Neuropathol. 2019 Jul 5. doi: 10.1007/s00401-019-02039-3. [Epubahead of print]